

白玉课题组发展深度有机质谱流式方法在单细胞水平揭示3-羟基丁酸的丰度异质性
单细胞代谢组学分析可以系统地“解读”细胞的生理和功能状态,对于揭示细胞表型异质性和阐明生物过程的分子机制至关重要。然而,由于现有单细胞代谢组学方法存在鉴定深度及其有限,单个细胞流式在线分析时间短导致无法获得二级质谱信息,且不具备同分异构体区分能力等诸多难题,单细胞代谢组学研究从方法到应用均面临巨大挑战。针对上述挑战,北京分子科学国家研究中心白玉课题组发展了深度有机质谱流式分析平台,可实现在线高效的细胞裂解以及高分辨质谱分析,单个细胞分析时间较现有技术延长40倍,可获得丰富的用于代谢物鉴定的谱图,单细胞代谢物鉴定数目可达600种,实现了普适性的单细胞异构体定性定量分析(图1),并揭示了MCF-7乳腺癌细胞存在的精细亚型。研究成果以“In-depth organic mass cytometry revealsdifferential contents of 3-hydroxybutanoicacid at the single-cell level”为题,于2024年5月23日发表在《自然-通讯》(Nature Communications)期刊上(Nature Commun. 2024,15,4387)。
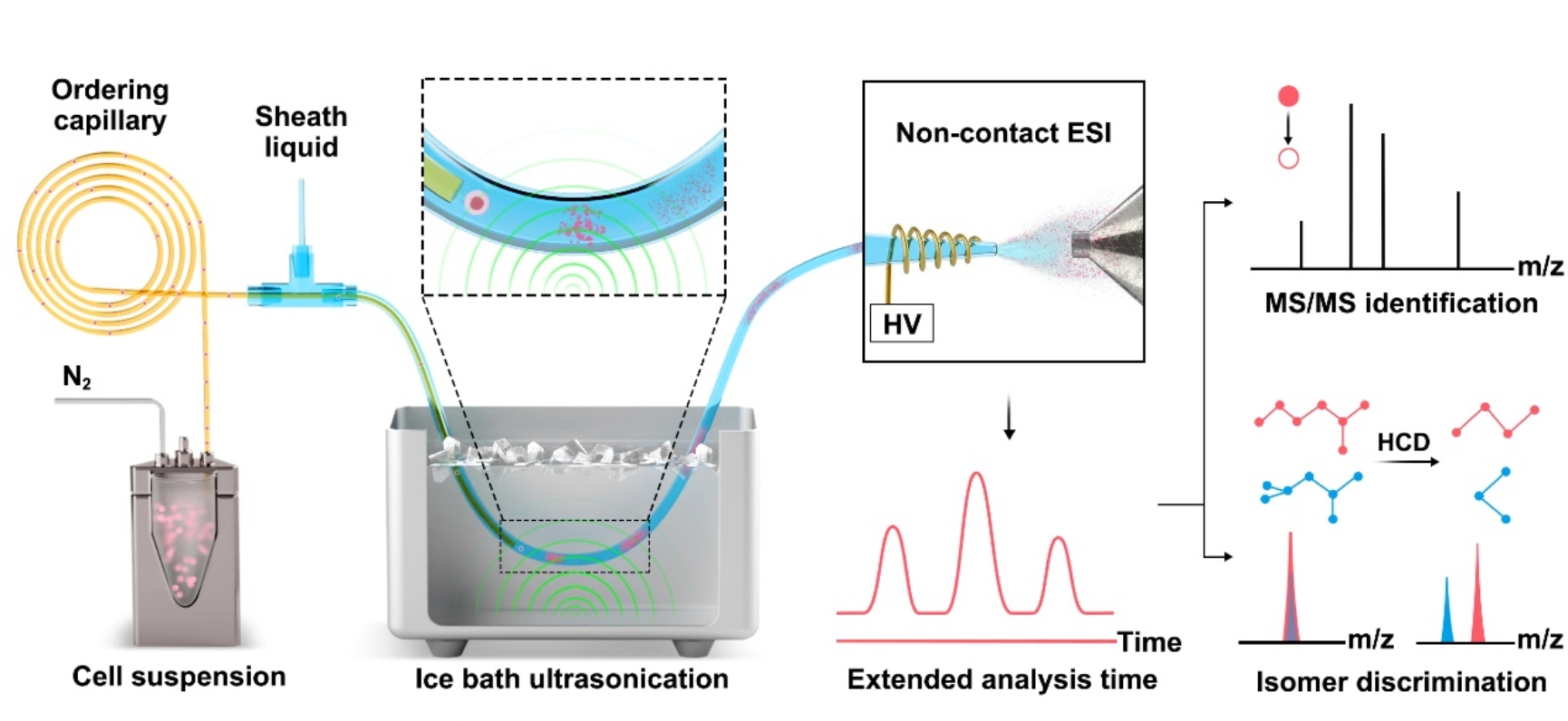
图1. 深度有机质谱流式分析平台的工作原理示意图
深度有机质谱流式分析平台由在线高效单细胞裂解装置和非接触电喷雾高分辨质谱组成。细胞通过迪恩流的流体作用在分离毛细管中实现单分散,鞘液毛细管通过T型三通的方式与分离毛细管同轴连接。细胞与鞘液接触后,通过在线冰浴超声促进细胞有效裂解和代谢物管内分散。在连续单细胞进样条件下,每个单细胞可以获得约25秒的平均分析时间,该时间内可有效采集高分辨率的一级以及二级质谱信息。大量的二级质谱数据也使得同分异构体鉴定和相对定量成为可能(图2)。
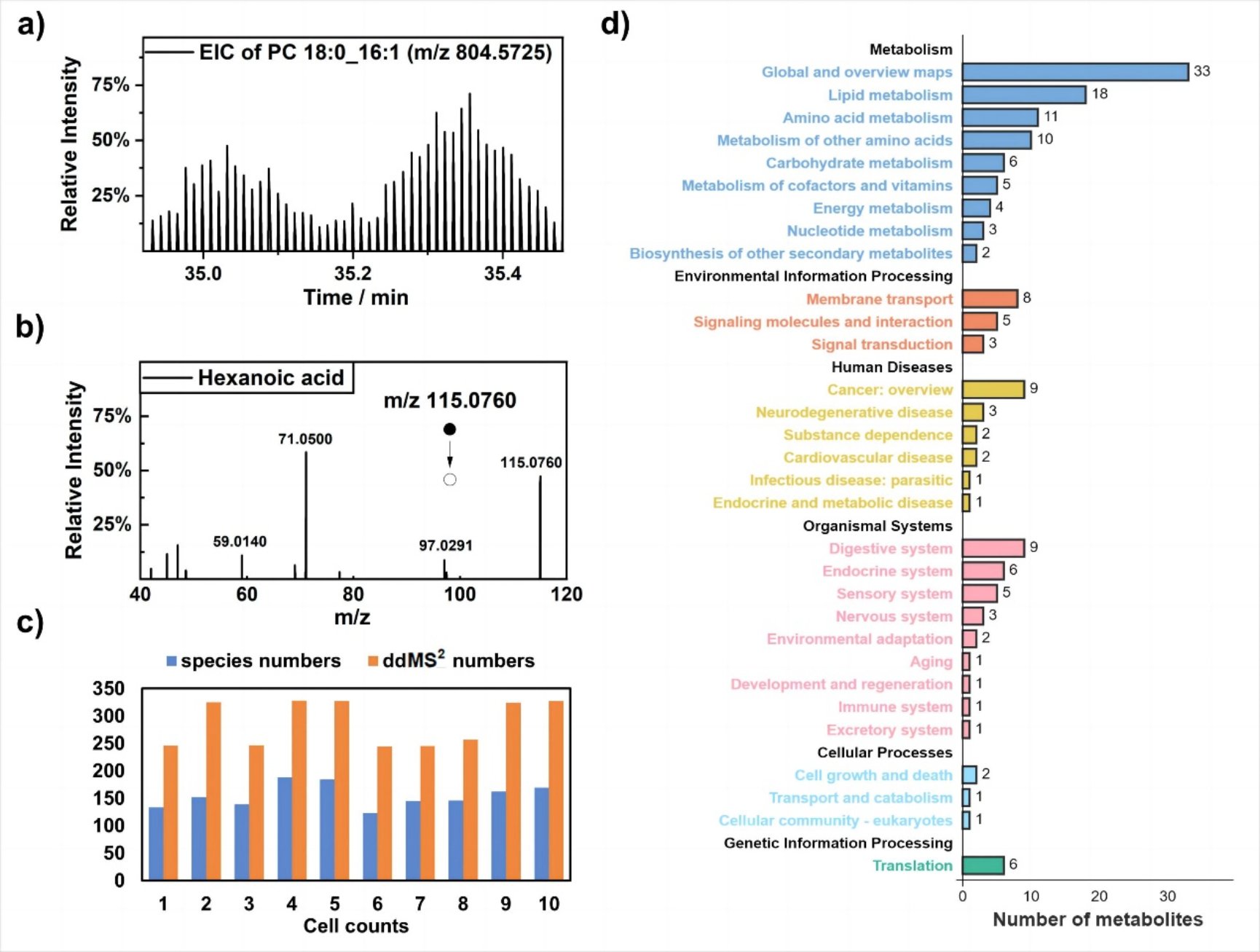
图2 基于二级质谱信息的单细胞代谢物深度分析
基于不同类型肿瘤细胞的代谢物信息证实了该流式平台具有普适性的细胞分型能力和异构体代谢物区分能力。利用3-羟基丁酸/4-羟基丁酸的异构体相对含量信息,通过UMAP可视化方法可成功实现MCF-7细胞的3种亚型精细区分。结合10×Genomics单细胞测序实验、蛋白质印迹实验以及荧光流式分选实验,该研究从转录组学水平以及蛋白表达水平均证实了3-羟基丁酸的丰度差异是导致肿瘤细胞分型的主要原因,且3-羟基丁酸与其下游多种抗氧化应激靶蛋白的表达量呈正相关,例如金属硫蛋白MT2A以及异质核糖核蛋白hnRNPA1,提示3-羟基丁酸的丰度差异性可能赋予肿瘤细胞不同的抵抗氧化应激环境的能力。该工作首次观察到MCF-7细胞可能存在潜在的细胞亚型,为深入理解肿瘤异质性以及乳腺癌精准治疗提供了新思路。
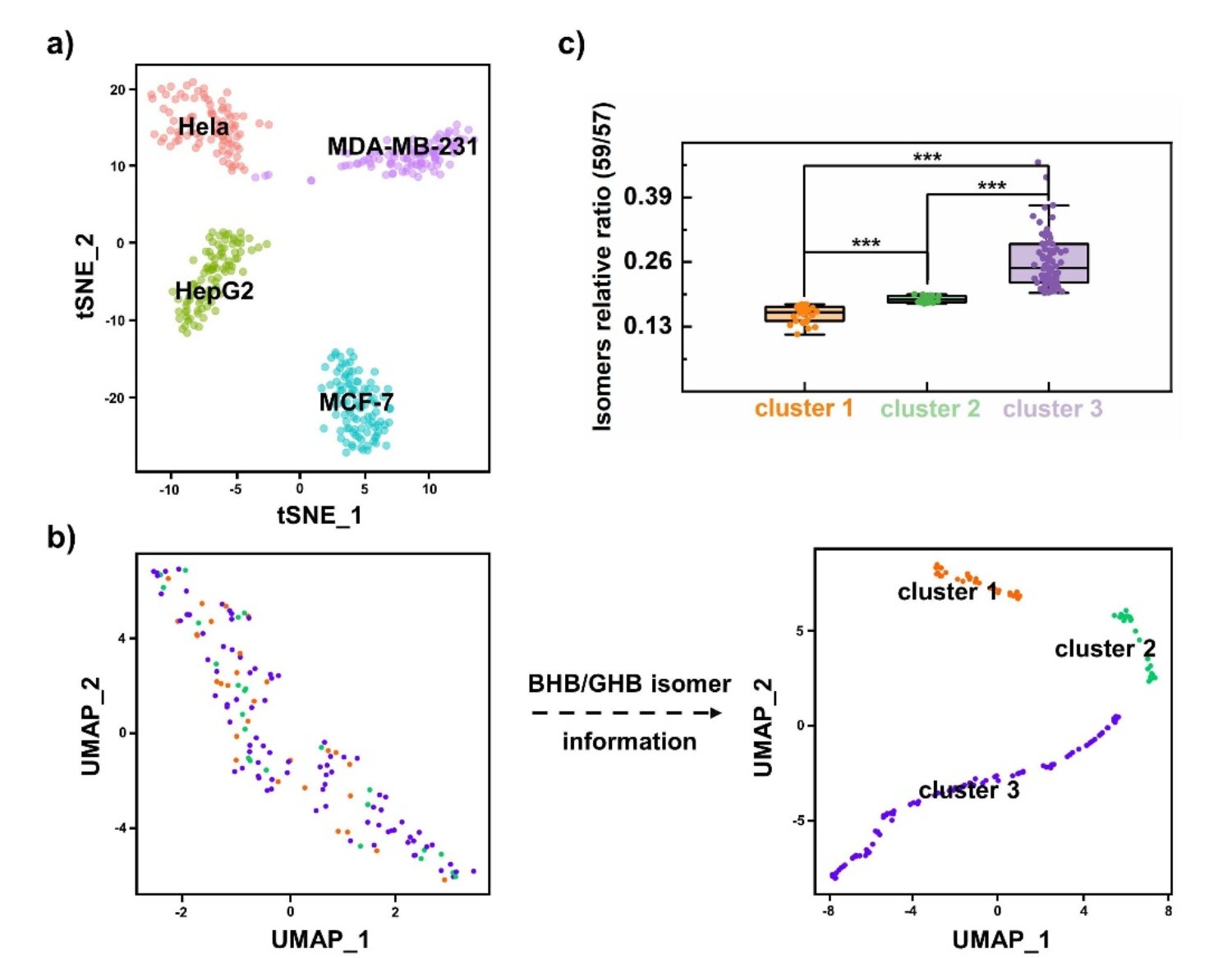
图3. 基于代谢物和异构体的肿瘤细胞可视化聚类和精细分型
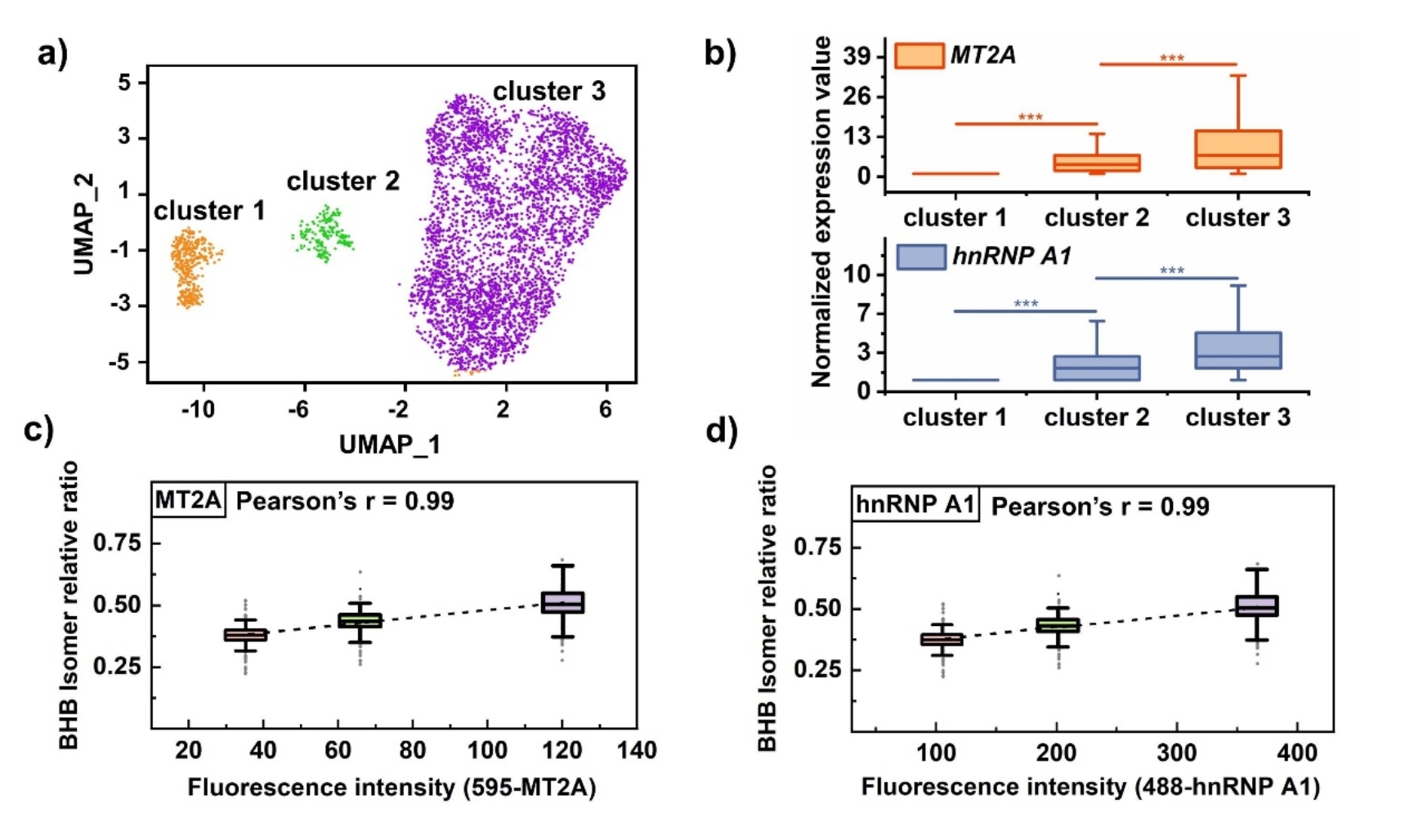
图4. 10×Genomics单细胞转录组测序MCF-7细胞的UMAP可视化结果、不同细胞簇间MT2A和hnRNP A1的归一化表达水平以及BHB相对丰度上述基因表达水平的变化
北京分子科学国家研究中心白玉教授是该论文的通讯作者,北京大学化学与分子工程学院博士研究生秦少杰是该论文的第一作者。该研究成果得到国家自然科学基金委、科技部和北京分子科学国家研究中心等机构的资助。研究工作中测序数据分析得到北京大学生物医学前沿创新中心文路副研究员、卢健森同学以及于红敏同学的大力支持,该研究还得到国家蛋白质科学研究(北京)北大分中心质谱平台刘栋老师的大力帮助。
原文链接:https://www.nature.com/articles/s41467-024-48865-2