

陈鹏/樊新元团队报道用于原代生物样品研究的生物正交光催化邻近标记技术CAT-S

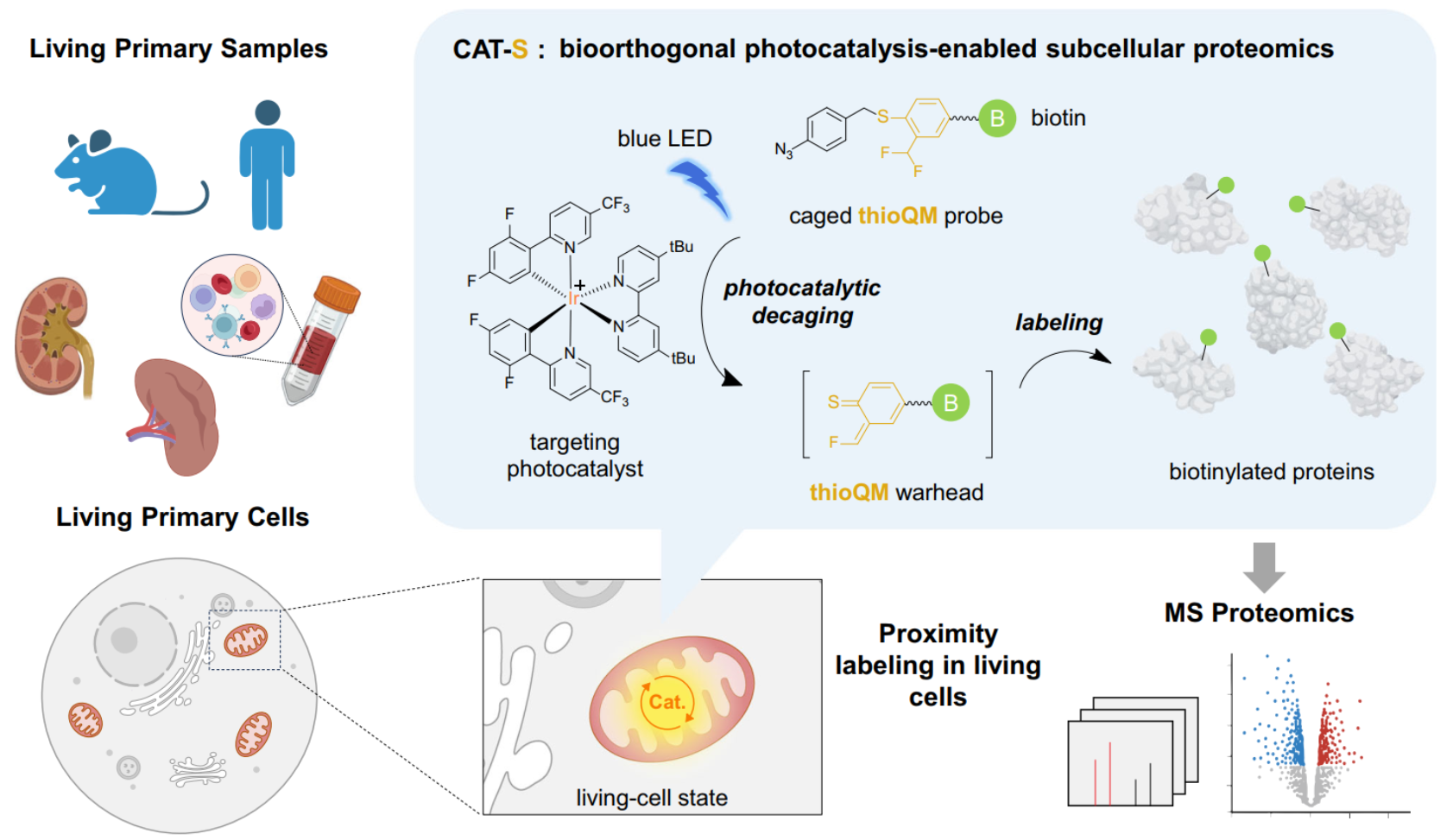
然而,组织原代、甚至人体临床等更为复杂样品的原位标记由于存在技术瓶颈而一直处于空白。2024年3月28日,北京分子科学国家研究中心陈鹏/樊新元团队在Nature Communications杂志发表了题为Bioorthogonal photocatalytic proximity labeling in primary living samples的研究论文。他们在前期开发的线粒体靶向的光催化邻近标记技术CAT-Prox的基础上,通过化学改造开发出新一代具有更高标记活性的硫代亚甲基醌探针,以适应更为复杂的原代组织的生物样品,从而实现了组织原代、甚至人体临床样品的线粒体蛋白质的原位标记(CAT-S技术)6。
作者首先基于前期的CAT-Prox技术进行系统性优化和升级,特别是针对传统常用的亚甲基醌(QM)标记探针进行化学改造,通过引入硫原子开发出新一代硫代QM标记基团(thioQM),在体外及活细胞内展现出比传统氧代QM显著提升的蛋白质标记效率。之后,在多种细胞系中验证了该方法对线粒体蛋白质的高效捕捉。通过与蛋白质谱技术的联用,能够在80%左右的高特异性水平下,定量鉴定300个以上的线粒体蛋白,描绘活细胞线粒体蛋白质组特征;通过综合分析在多种细胞系中获取的数据,进一步发掘并验证了PTPN1、SLC35A4 uORF及TRABD三个新的线粒体定位蛋白,展现该技术在发现新的线粒体蛋白方面的能力。

原代活细胞样品(尤其是临床样品)上的应用一直是邻近标记领域的难点。对此,研究者通过CAT-S技术实现了对小鼠肾脏、脾脏的解离细胞样品中线粒体蛋白的邻近标记捕捉,避免了酶法邻近标记技术对转基因动物模型的依赖。据此,研究者定量比较了肥胖诱导II型糖尿病小鼠和健康小鼠中的肾脏线粒体蛋白质组特征,发现一系列在疾病状态下表达变化的线粒体蛋白。其中,脂代谢相关酶类的变化较为显著,其中Aldh3a2、Acsm2的下降可能造成了相关脂质代谢物的累积,潜在地促进了糖尿病相关的肾病发展。此外,研究者还证明了CAT-S技术能够对人血液样品中原代T细胞进行原位线粒体蛋白标记,展现了在临床样品上的应用潜力。
综上,该工作对生物正交光催化标记化学进一步发展,实现了其在动物组织和临床来源的原代细胞的应用,并进行了原位线粒体蛋白质组解析。其无需基因改造、通用性、光控时空分辨等特点,为原代样品的亚细胞蛋白质组学研究开辟了新途径。基于该策略,进一步发展靶向其他亚细胞区域的生物正交标记方法、探究更多疾病相关的组学信息,将是未来值得期待的方向。
北京分子科学国家研究中心樊新元和陈鹏教授为本文的通讯作者,博士后刘子琦与博士研究生郭福虎为本文的共同第一作者。该工作得到来自国家自然科学基金、科技部重点研发计划、北京市自然科学基金、李革-赵宁生命科学青年研究基金等项目经费的支持。
原文链接:https://www.nature.com/articles/s41467-024-46985-3
参考文献:
1.Geri, J. B.; Oakley, J. V.; Reyes-Robles, T.; Wang, T.; McCarver, S. J.; White, C. H.; Rodriguez-Rivera, F. P.; Parker, D. L.; Hett, E. C.; Fadeyi, O. O.; Oslund, R. C.; MacMillan, D. W. C., Microenvironment mapping via Dexter energy transfer on immune cells. Science 2020,367 (6482), 1091.
2.Seath, C. P.; Burton, A. J.; Sun, X.; Lee, G.; Kleiner, R. E.; MacMillan, D. W. C.; Muir, T. W., Tracking chromatin state changes using nanoscale photo-proximity labelling. Nature 2023, 616 (7957), 574-580.
3.Huang, Z.; Liu, Z.; Xie, X.; Zeng, R.; Chen, Z.; Kong, L.; Fan, X.; Chen, P. R., Bioorthogonal Photocatalytic Decaging-Enabled Mitochondrial Proteomics. J. Am. Chem. Soc. 2021,143 (44), 18714-18720.
4.Liu, Z.; Xie, X.; Huang, Z.; Lin, F.; Liu, S.; Chen, Z.; Qin, S.; Fan, X.; Chen, P. R., Spatially resolved cell tagging and surfaceome labeling via targeted photocatalytic decaging. Chem 2022, 8 (8), 2179-2191.
5.Zhang, Y.; Liu, S.; Guo, F.; Qin, S.; Zhou, N.; Fan, X.; Chen, P. R., Bioorthogonal photocatalytic quinone methide decaging for cell-cell interaction labeling. bioRxiv 2023, 2023.04. 08.536099.
6.Liu, Z.; Guo, F.; Zhu, Y.; Qin, S.; Hou, Y.; Guo, H.; Lin, F.; Chen, P. R.; Fan, X., Bioorthogonal photocatalytic proximity labeling in primary living samples. Nat. Commun. 2024,15 (1), 2712.