

雷晓光团队揭示植物来源分子间Diels-Alder反应酶的自然演化奥秘
植物来源的天然产物通常是植物的次级代谢产物,这些次级代谢产物既是调控植物生长发育的信号分子,又可以通过与环境的相互作用来提高植物生存和抵抗外界压力的能力。植物之所以能够产生化学结构异常多样、丰富的次级代谢产物,是因为植物体内存在大量的催化反应酶,帮助快速、高效地合成相关有机小分子。因此,植物被认为是自然界中“最伟大的化学家”。但是,植物中生物合成酶究竟是如何通过自然演化来获得机制独特的催化活性一直以来都是没有被完整回答的重要科学问题。
Diels-Alder(D-A)反应,作为有机合成领域中重要的人名反应,一直以其独特的地位在天然产物的全合成中发挥着重要的作用。鉴于其广泛的应用前景和深远影响,从自然界中探寻能够催化D-A反应的酶(D-A酶)已成为生物合成领域的研究焦点之一。在当前的生物合成领域中,研究者们正致力于挖掘自然界中潜藏的D-A酶资源,以期通过酶催化手段实现D-A反应的高效、绿色合成。
在目前已知的D-A酶中,不同功能的D-A酶在序列和结构上缺乏明显的保守性。这一特性使得新颖D-A酶的鉴定工作变得尤为困难,特别是对于那些源自植物的D-A酶而言,其鉴定难度更是成倍增加。因此,对D-A酶演化机制的深入研究显得尤为迫切,这将有望揭开自然界“创造”这类酶的神秘面纱。
在实验室的前期探索中,北京分子科学国家研究中心雷晓光课题组首次在非模式植物白桑中鉴别出两种具有不同endo/exo选择性的分子间D-A酶,并对这些酶的催化机制进行了详尽而系统的研究,相关成果已相继发表在《自然·化学》和《自然·催化》等期刊上(Nature Chemistry 2020, 12, 620–628; Nature Catalysis 2021, 4, 1059–1069)。基于这些前期的研究,该团队进一步观察到,来源于桑科植物的分子间D-A酶MaDA与二烯体合成酶MaMO在序列和结构上展现出了显著的相似性,两者均归属于FAD偶联氧化酶家族中的类BBE酶亚家族。然而,令人惊奇的是,尽管大多数类BBE家族的酶主要催化氧化反应,但D-A酶却催化非氧化反应,这一特性暗示MaDA酶很可能是该家族中一个具有新功能化的成员。这点引发了研究者的浓厚兴趣。
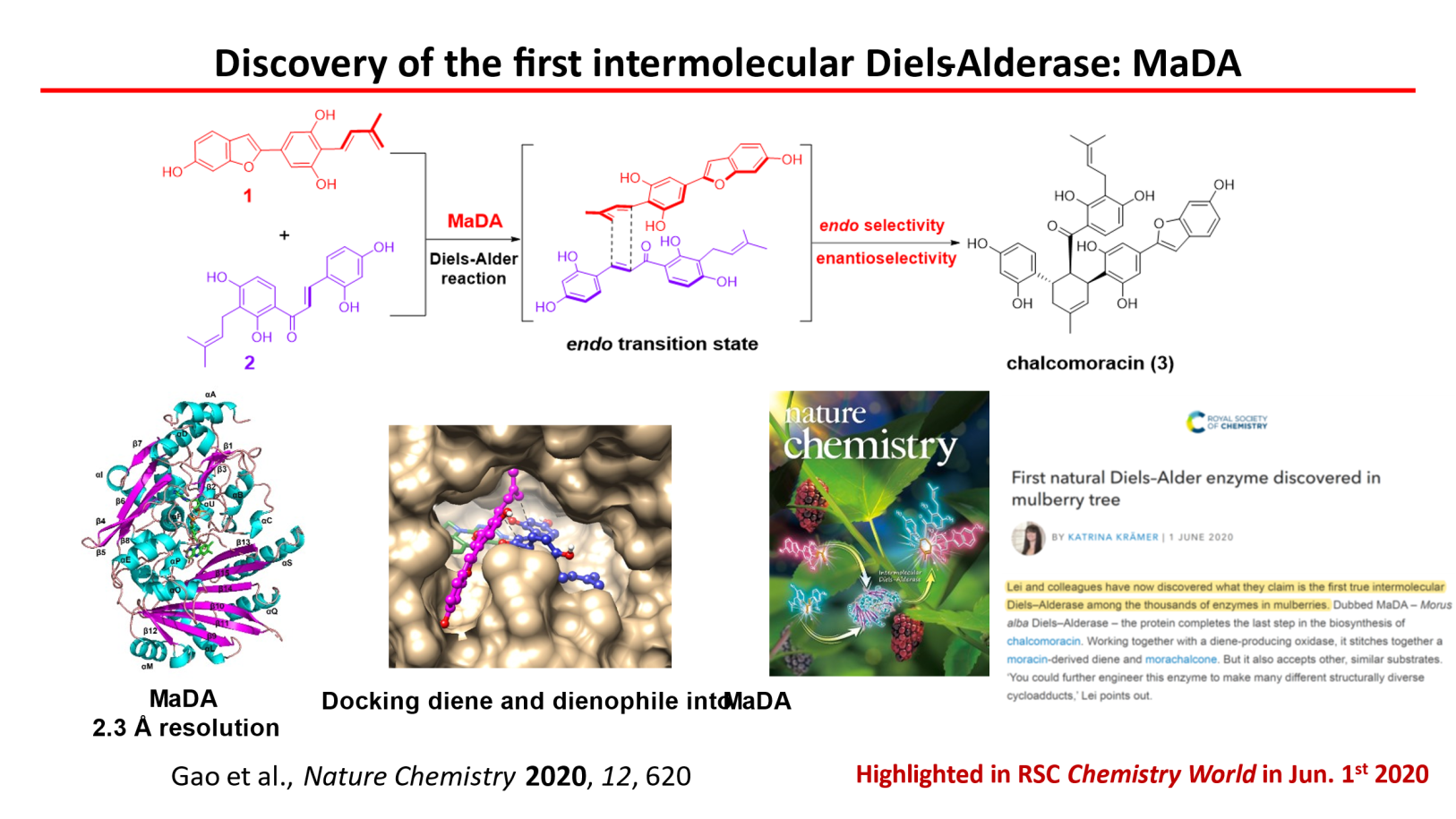
图1,首例植物来源分子间D-A反应酶的发现
为深入探索D-A酶的自然演化起源和机制,雷晓光教授团队采用了一系列技术手段,包括祖先序列重建、生物信息学挖掘、结构生物学、酶学、分子动力学模拟以及定点诱变实验等,全面而系统地揭示了桑树中FAD依赖的氧化环化酶演化为D-A酶和其功能相关的二烯体合成酶的历程。研究团队不仅阐明了这一演化过程中的具体机制,还鉴定了参与功能转变的关键的残基突变,同时揭示了酶功能转变过程中底物的微妙调整以适应功能转变的可能机制。
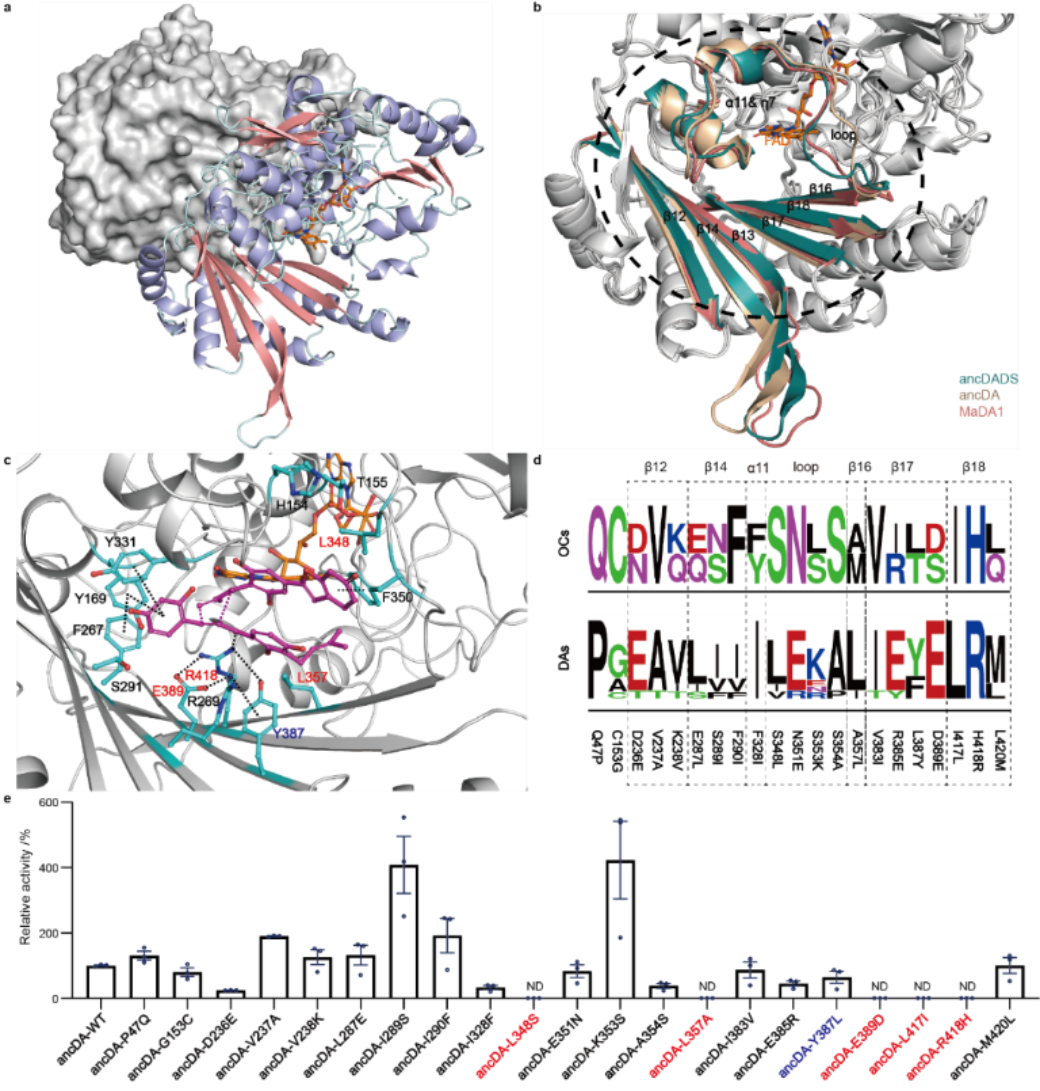
图2,D-A酶的结构、活性口袋和关键突变残基
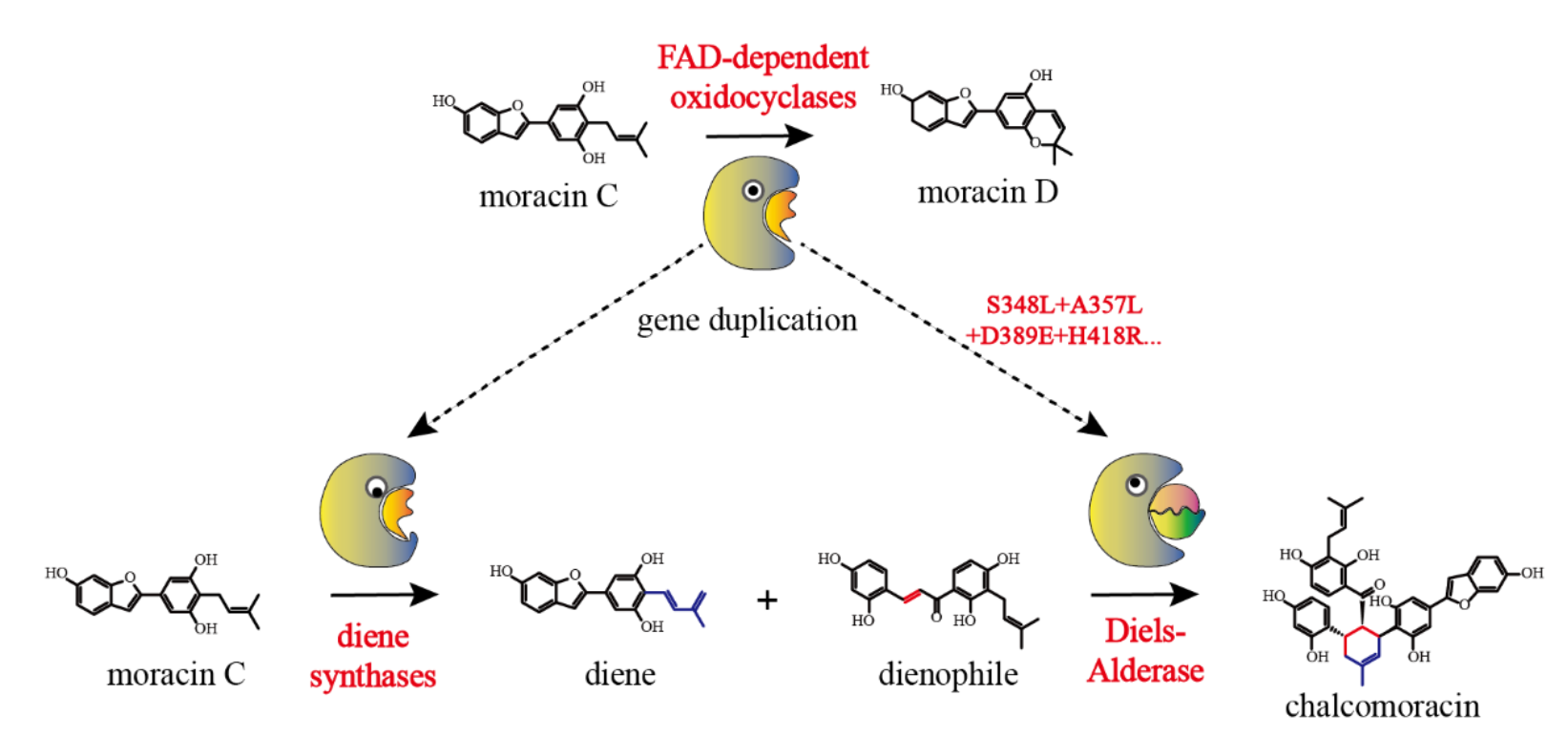
图3,FAD依赖的氧化环化酶演化为D-A酶的模型
该工作以“The evolutionary origin of naturally occurring intermolecular Diels-Alderases from Morus alba”为题,在《Nature Communications》期刊上发表(DOI: 10.1038/s41467-024-46845-0)。自1928年Diels-Alder反应被首次报道以来,尽管众多科学家在过去的几十年里致力于对该反应的生物合成酶进行深入的挖掘和鉴定,但所发现的D-A酶数量仍然寥寥无几。这项关于D-A酶演化机制的重要研究为未来D-A酶的挖掘和改造提供了新的思路和方向,为生物合成和化学合成领域的发展注入新的活力。同时该研究也为深入理解植物天然产物的生物合成途径与自然进化提供了重要思路。

北京京分子科学国家研究中心、北大-清华生命科学联合中心雷晓光教授和团队中高磊特聘副研究员为论文的共同通讯作者。雷晓光课题组博士生丁琪和郭念昕为共同第一作者;范俊萍特聘副研究员、吴东山、杨军以及美国东北大学翁经科教授和其研究生Michelle McKee也参与了相关研究工作。相关生物信息学分析和计算化学在北京大学高性能计算平台上完成。该研究得到了国家重点研发计划、国家自然科学基金委员会、北京分子科学国家研究中心、北大-清华生命科学联合研究中心、新基石基金会等项目和单位的资助。