

陈鹏/樊新元团队与合作者开发生物正交“色氨酸脱笼”技术助力上万潜在蛋白的特异激活
因其独特的结构,色氨酸是蛋白质中介导分子相互作用类型最多、也最“昂贵”的天然氨基酸,其生物合成的“成本”远超其他天然氨基酸,且只由UGG一个密码子编码。【1】由于色氨酸的独特性和稀缺性,色氨酸往往出现在蛋白质的关键位点,稳定蛋白质结构、调节蛋白质构象、调控蛋白质相互作用、参与分子识别和催化等重要过程。【2-3】如果能够在活细胞内精准操纵特定色氨酸的功能及相互作用,就有望获得一种特异激活含色氨酸蛋白质的通用技术。然而目前尚无对色氨酸进行“可逆化学编辑”的相关报道。
2024年2月28日,北京分子科学国家研究中心陈鹏/樊新元团队与浙江大学林世贤团队合作,在Nature Chemistry杂志在线发表了题为“Genetically encoded bioorthogonal tryptophan decaging in living cells”的研究论文。该研究开发了一种能在活细胞中“笼锁”和“脱笼解锁”蛋白质中任意色氨酸功能的通用方法(Trp-CAGE,图1)。该方法利用基因密码子扩展技术将“笼锁”色氨酸(Caged Tryptophan)引入目标蛋白的特定位点,实现其功能的暂时屏蔽;然后,利用新发展的生物正交剪切反应在活细胞中完成可控“脱笼”,实现各类蛋白质家族的精准“激活”和功能解析。通过计算机模型预测,该“色氨酸脱笼”技术可对超过28000个来自不同物种的候选蛋白质进行功能获得性研究。

陈鹏课题组长期致力于发展适用于活细胞的生物正交反应,在国际上率先提出并发展了生物正交剪切反应【4】,即通过对目标蛋白关键残基的保护-脱保护,实现对其活性的原位关-开”调控(即笼锁-脱笼,caging-decaging)。这种方法普适性广,对蛋白结构改动小,可以最大程度还原天然蛋白的活性。在前期的研究工作中,陈鹏课题组将生物正交剪切反应运用于多种氨基酸残基的脱笼,在活细胞中实现了不同种类酶活性的原位、瞬时激活【5-8】。“色氨酸脱笼”是其课题组在生物正交剪切反应领域的全新突破!
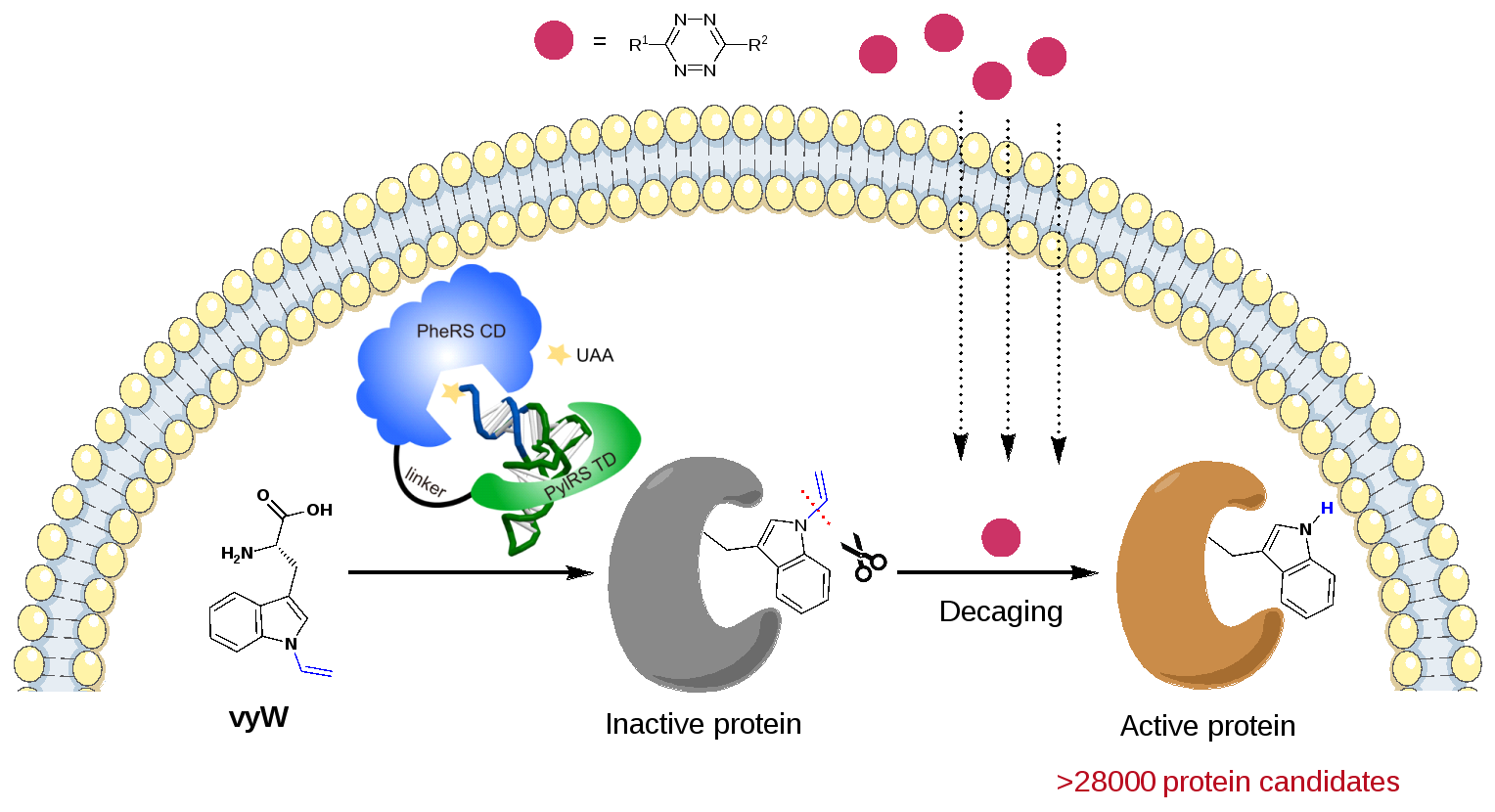
图1. 色氨酸“笼锁-脱笼”(Trp-CAGE)策略的示意图
在研究中,研究人员首先设计了能掩蔽几乎所有色氨酸相互作用的非天然氨基酸,并发展了与之配套的生物正交剪切反应。研究人员在色氨酸的吲哚氮原子上引入一个额外的π系统(乙烯基),通过共轭效应削弱了吲哚环的π能量,从而同时阻断了色氨酸的极性相互作用、疏水相互作用和π相互作用(图2)。此外,研究人员还注意到乙烯基的HOMO轨道能量因共轭作用而上升,使其参与逆电子需求的狄尔斯-阿尔德反应(IEDDA)的活性增强。基于这一发现,研究人员开发了首个针对吲哚类结构的IEDDA-剪切反应。该反应可在PBS溶液中稳定进行,能够在30分钟内以大于80%的收率得到脱笼产物。
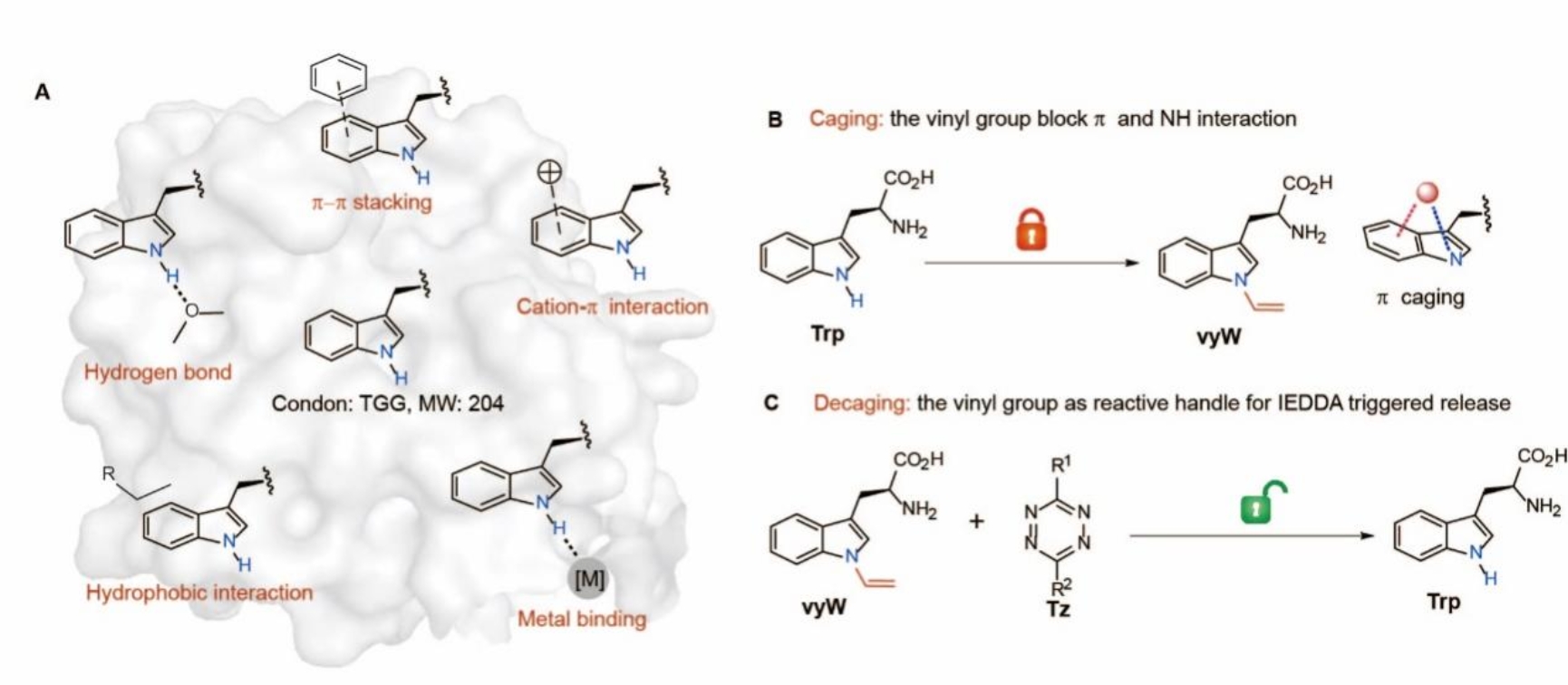
图2. 非天然氨基酸的设计及生物正交剪切反应的发展
接下来,研究人员采用了遗传密码子扩展策略以位点特异性的方式将“笼锁”色氨酸(N-乙烯基色氨酸,vyW)引入蛋白质中,并采用了“级联进化”的策略对嵌合体苯丙氨酸氨酰-tRNA合成酶(chPheRS)加以进化筛选【9】。首先,他们选择了一种与vyW化学结构相似且只相差一个碳原子的过渡化合物N-甲基色氨酸(1MW)。通过定向进化筛选,成功获得了能够识别1MW的突变体(1MWRS),并建立了1MW和vyW与这些突变体之间的对接模型。基于对接模型的不同,研究人员有针对性地对1MWRS进行了三个关键残基的突变,并最终成功鉴定出一种高效准确识别vyW的氨酰-tRNA合成酶(vyWRS)(图3)。随后,研究者们利用模型蛋白质在体外以及活细胞中评估了脱笼反应的效率。实验结果显示,对于体外纯化的模型蛋白质-GFP上的vyW,其脱笼效率可超过>90%,而在活细胞中,使用Renilla Luciferase (RLuc)报告系统评估的脱笼效率达到了54%。

图3. 氨酰-tRNA的进化及Trp-CAGE在体外及活细胞中的验证
在此基础上,研究人员建立了蛋白质激活的通用平台。通过在PDB数据库中使用新建的算法来搜索含有关键色氨酸的蛋白质,发现超过28,000个蛋白质的色氨酸-配体距离在5Å以内。这表明这些蛋白质可以通过色氨酸脱笼技术进行功能研究。色氨酸残基在蛋白质中的化学相互作用可以归类为五种主要类型,包括氢键作用、疏水相互作用、金属结合、π-π堆积和阳离子-π相互作用。研究人员针对每种相互作用挑选了示例蛋白,并成功使用Trp-CAGE策略“沉默”和“恢复”了相应的蛋白质功能。Trp-CAGE策略的应用范围广泛,可以精确调控多种蛋白质的生理过程,包括别构作用、蛋白质成熟过程、酶活口袋调控,蛋白质-RNA相互作用以及蛋白质-蛋白质相互作用等。该策略适用于各种蛋白质家族,包括荧光蛋白(用于生物标记和显微镜研究)、金属结合蛋白(参与金属离子的配位和催化反应)、激酶(调控信号传导通路)、荧光素酶(用于生物发光实验)、翻译起始因子(调节蛋白质合成)和翻译后修饰读取蛋白(参与蛋白质修饰)等。通过Trp-CAGE策略,这些蛋白质的功能可以被精准的调控和研究(图4)。

图4. 基于Trp-CAGE策略的通用型蛋白质激活平台
研究人员进一步关注了利用Trp-CAGE来调控表观遗传“阅读器”蛋白与组蛋白的相互作用。从PDB数据挖掘中,研究人员分别确定了27个、134个和133个含有赖氨酸单甲基化、双甲基化和三甲基化的识别结构域中含有关键的色氨酸。这些结构域可以识别组蛋白H3上K4、K9、K27和K36位点的赖氨酸甲基化。【10】研究证实,Trp-CAGE策略可以在体外以及活细胞中调控所有选定的阅读结构域(包括PHD、Chromo、BAH和Tudor结构域)与组蛋白H3上的四个甲基化赖氨酸位点(K4、K9、K27和K36)之间的相互作用(图5)。
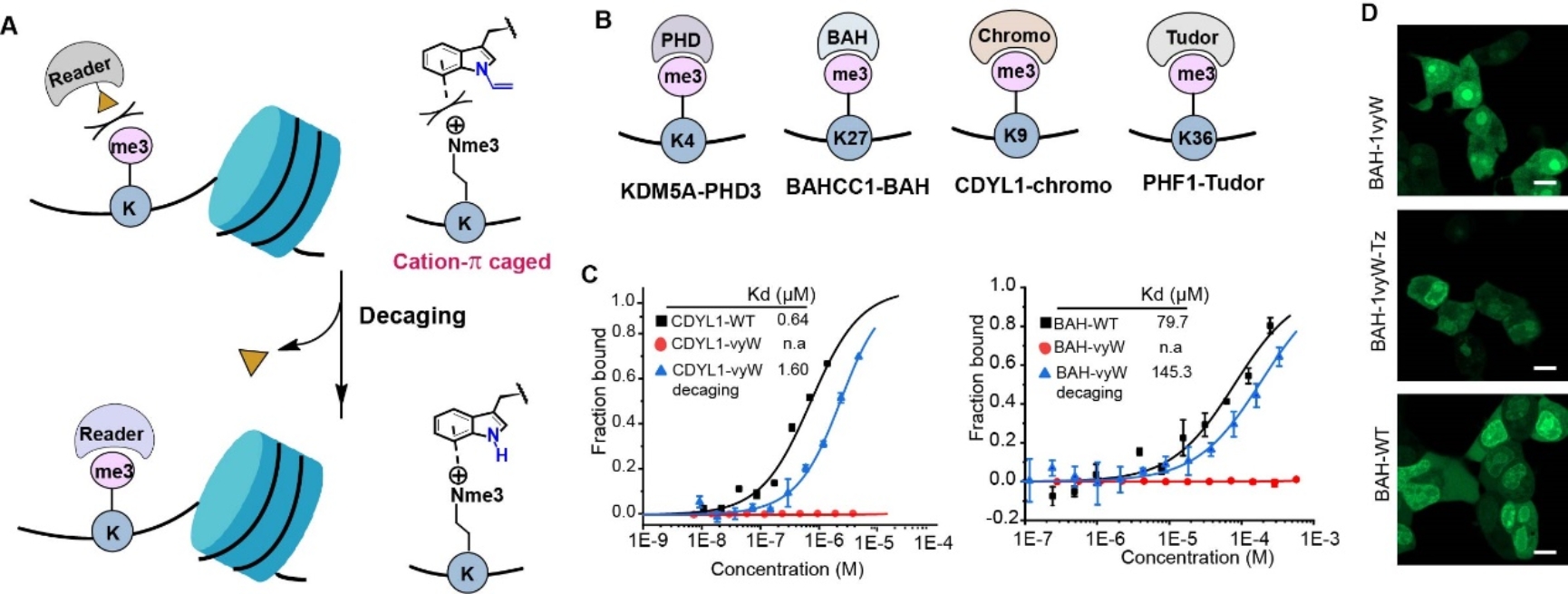
图5. 在活细胞中调控表观遗传读码蛋白
综上,该工作发展了一种独特且通用的色氨酸脱笼方法,用于“化学”激活蛋白质特定位点上的色氨酸,以实现相应蛋白质的功能获得性研究。该研究以共轭化学作为设计思路,使用乙烯基作为保护基团,通过共轭吲哚的π系统成功地阻断了色氨酸的几乎所有相互作用类型。这种独特的保护基团可以通过生物正交剪切反应迅速脱除,实现对活细胞内蛋白质的实时、原位激活。更令人兴奋的是,该策略具有广泛的适用性,可以操纵各种不同类型的蛋白质,包括荧光蛋白、金属结合蛋白、激酶、荧光素酶、翻译起始因子和组蛋白翻译后修饰阅读蛋白等。根据计算预测,该策略可以在超过28,000个候选蛋白质上进行生物正交激活调控,展示了其巨大的应用潜力。这一突破为探索和理解蛋白质功能提供了全新的工具。
北京分子科学国家研究中心陈鹏/樊新元课题组博士后朱玉超、浙江大学林世贤课题组博士后丁文龙为论文的共同第一作者,陈鹏教授、林世贤研究员和樊新元副研究员为论文的共同通讯作者。该研究获得了国家重点研发计划、国家自然科学基金、北京市自然科学基金、北京分子科学国家研究中心、新基石研究员计划、生物医学峰基金以及博士后创新人才支持计划的资助。
文章链接:https://www.nature.com/articles/s41557-024-01463-7
参考文献:
1. Barik, S. The Uniqueness of Tryptophan in Biology: Properties, Metabolism, Interactions and Localization in Proteins. Int. J. Mol. Sci.21, 8776, (2020).
2. Cuello, L. G. et al. Structural basis for the coupling between activation and inactivation gates in K+ channels. Nature 466, 272-275, (2010).
3. Xue, Y. et al. Cu(I) recognition via cation-π and methionine interactions in CusF. Nat. Chem. Biol. 4, 107-109, (2007).
4. Li, J. & Chen, P. R. Development and application of bond cleavage reactions in bioorthogonal chemistry. Nat. Chem. Biol. 12, 129-137, (2016)
5. Wang, J., Wang, X., Fan, X. & Chen, P. R. Unleashing the Power of Bond Cleavage Chemistry in Living Systems. ACS. Cent. Sci. 7, 929-943, (2021).
6. Wang, J. et al. Time-resolved protein activation by proximal decaging in living systems. Nature 569, 509-513, (2019)
7. Li, J., Jia, S. & Chen, P. R. Diels-Alder reaction-triggered bioorthogonal protein decaging in living cells. Nat. Chem. Biol. 10, 1003-1005, (2014)
8. Li, J. et al. Palladium-triggered deprotection chemistry for protein activation in living cells. Nat. Chem. 6, 352-361, (2014).
9. Ding, W. et al. Chimeric design of pyrrolysyl-tRNA synthetase/tRNA pairs and canonical synthetase/tRNA pairs for genetic code expansion. Nat. Commun.11, 3154, (2020).
10. Zhao, H. et al. Manipulating Cation-π Interactions with Genetically Encoded Tryptophan Derivatives. J. Am. Chem. Soc.144, 6742-6748, (2022).